คริสเปอร์

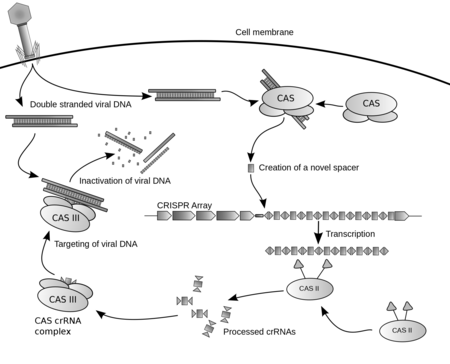
คริสเปอร์ (อังกฤษ: CRISPR) เป็นวงศ์ของลำดับดีเอ็นเอในแบคทีเรีย ซึ่งในลำดับมีส่วนของดีเอ็นเอ (DNA snippets) จากไวรัสที่เคยโจมตีแบคทีเรียนั้น แบคทีเรียใช้ส่วนเหล่านี้ในการตรวจหาและทำลายดีเอ็นเอจากไวรัสที่คล้ายกันในการโจมตีครั้งถัดไป ลำดับเหล่านี้ทำหน้าที่สำคัญในระบบภูมิคุ้มกันของแบคทีเรีย[3] และเป็นพื้นฐานของเทคโนโลยีที่เรียกว่า คริสเปอร์/แคสไนน์ (อังกฤษ: CRISPR/Cas9) ที่สามารถใช้เปลี่ยนยีนภายในสิ่งมีชีวิต[4]
ระบบคริสเปอร์/แคสเป็นระบบภูมิคุ้มกันโพรแคริโอตที่ช่วยในการต่อต้านส่วนประกอบทางพันธุกรรมแปลกปลอม เช่นเดียวกับที่อยู่ในพลาสมิด (plasmid) และ เฟจ (phages)[5][6][7] ซึ่งเป็นภูมิคุ้มกันที่เกิดขึ้นภายหลัง (acquired immunity) อาร์เอ็นที่มีลำดับสเปสเซอร์ (spacer sequence) ช่วยโปรตีนแคส (CRISPR-associated, Cas) ในการจำแนกและตัดดีเอ็นเอจากภายนอก ขณะที่โปรตีนแคสอื่นที่นำโดยอาร์เอ็นเอตัดอาร์เอ็นเอจากภายนอก[8] คริสเปอร์ถูกพบในประมาณ 40% ของจีโนมแบคทีเรียและใน 90% ของอาร์เคียที่ถูกหาลำดับดีเอ็นเอแล้ว[9]
CRISPR ย่อมาจาก Clustered Regularly Interspaced Short Palindromic Repeats[10] ชื่อถูกตั้งก่อนจะรู้ที่มาและหน้าที่ของลำดับย่อยอินเตอร์สเปส (interspacing subsequence) ณ ตอนนั้น คริสเปอร์ถูกมองว่าเป็นส่วนหนึ่งของดีเอ็นเอโพรแคริโอตที่มีลำดับเบสสั้น ๆ ที่เรียงตัวซ้ำ ๆ ลำดับซ้ำพาลินโดรม (palindromic repeat) เป็นลำดับนิวคลีโอไทด์ที่เหมือนกันเมื่ออ่านจากทั้งสองด้าน การซ้ำแต่ละครั้งตามด้วยดีเอ็นเอสเปสเซอร์จากการพบกับดีเอ็นเอภายนอกครั้งก่อน (เช่น ไวรัส หรือ พลาสมิด)[11] ต่อจากลำดับคริสเปอร์ก็มีกลุ่มเล็ก ๆ ของยีนแคส (CRISPR-associated system, cas)
ระบบคริสเปอร์/แคสแบบง่ายที่เรียกว่าคริสเปอร์/แคสไนน์ถูกปรับแต่งเพื่อแก้ไขจีโนม จีโนมสามารถถูกตัดในตำแหน่งที่ต้องการ โดยการส่งแคสไนน์นิวคลีเอส (Cas9 nuclease) ประกอบกับไกด์อาร์เอ็นเอ (guide RNA, gRNA) เข้าไปในเซลล์ ทำให้สามารถกำจัดยีนที่มีอยู่หรือเพิ่มยีนใหม่เข้าไปได้[12][13][13][14] กลุ่มรวม Cas9-gRNA ทำหน้าที่เหมือน กลุ่มรวม CAS III crRNA บนแผนภาพด้านข้าง
เทคนิคการแก้ไขจีโนมคริสเปอร์/แคสมีศักยภาพในการนำไปประยุกต์ใช้ในหลายด้าน รวมถึงทางการแพทย์ และทางการปรุงแต่งเมล็ดพืชทางเกษตร การใช้กลุ่มรวม CRISPR/Cas9-gRNA เพื่อแก้ไขจีโจม[15][16] ถูกเลือกโดยสมาคมเพื่อความก้าวหน้าทางวิทยาศาสตร์อเมริกัน (AAA) ให้เป็นการค้นพบครั้งใหญ่ใน พ.ศ. 2558[17] ข้อกังวลด้านชีวจริยธรรมถูกกล่าวถึงเกี่ยวกับความเป็นไปได้ในการใช้เทคนิคนี้เพื่อแก้ไขเซลล์สืบพันธุ์[18]
ประวัติ
[แก้]การค้นพบของกลุ่มดีเอ็นเอลำดับซ้ำเกิดขึ้นโดยอิสระในสามส่วนของโลก หนึ่งในการค้นพบครั้งแรกเกิดขึ้นเมื่อ พ.ศ. 2530 ณ มหาวิทยาลัยโอซะกะในประเทศญี่ปุ่น นักวิจัย โยชิซุมิ อิชิโนะ (Yoshizumi Ishino) และคณะเผยแพร่ผลการทดลองเกี่ยวกับลำดับของยีนชื่อว่า "iap" และความเกี่ยวข้องกับ E. coli ความก้าวหน้าทางเทคโนโลยีในช่วงคริสต์ทศวรรษ 1990 ทำให้พวกเขาสามารถดำเนินงานวิจัยต่อและเพิ่มความเร็วของการหาลำดับด้วยเทคนิคที่เรียกว่าเมตาจีโนมิกส์ (metagenomics) พวกเขาสามารถเก็บตัวอย่างน้ำทะเลหรือดินและหาลำดับดีเอ็นเอในตัวอย่างนั้น
กลไก
[แก้]



ภูมิคุ้มกันคริสเปอร์-แคสเป็นกระบวนการทางธรรมชาติของแบคทีเรียและอาร์เคีย คริสเปอร์-แคสป้องกันการติดเชื้อเฟจ, คอนจูเกชัน (conjugation) และการแปลงพันธุ์ทางธรรมชาติ โดยการทำลายกรดนิวคลีอิกจากภายนอกที่เข้ามาในเซลล์[19]
การได้มาซึ่งสเปสเซอร์
[แก้]เมื่อจุลินทรีย์ถูกบุกรุกโดยไวรัส ขั้นแรกของการตอบสนองของภูมิคุ้มกันคือการจับดีเอ็นเอของไวรัสและใส่เข้าไปในโลคัสคริสเปอร์ในรูปแบบของสเปสเซอร์ การที่ Cas1 และ Cas2 อยู่ในระบบภูมิคุ้มกันคริสเปอร์-แคสทั้งสองแบบ ชี้ว่าพวกมันมีส่วนในการได้มาซึ่งสเปสเซอร์ งานวิจัยการกลายพันธุ์ยืนยันสมมติฐานนี้ และแสดงว่าการนำยีน cas1 หรือ cas2 ออกทำให้ไม่สามารถจัดหาสเปสเซอร์ได้ ในขณะที่ไม่ส่งผลกระทบต่อการตอบสนองของภูมิคุ้มกันคริสเปอร์[20][21][22][23][24]
โปรตีน Cas1 หลายโปรตีนถูกศึกษาลักษณะเฉพาะและหาโครงสร้าง[25][26][27] โปรตีน Cas1 มีลำดับกรดอะมิโนหลากหลาย อย่างไรก็ตาม โครงสร้างผลึกมีลักษณะคล้ายกัน และโปรตีน Cas1 ที่ถูกทำให้บริสุทธิ์ทั้งหมดเป็นนิวคลีเอส/อินทีเกรสที่ต้องพึ่งธาตุโลหะ และจับกับดีเอ็นเอโดยไม่พึ่งกับลำดับ[28] โปรตีน Cas2 ที่ถูกเลือกได้รับการศึกษาลักษณะเฉพาะ และพบว่าพวกมันแสดงกิจกรรมเอนโดริโบนิวคลีเอสเฉพาะต่อ ssRNA (สายเดี่ยว)[29] หรือ dsDNA (สายคู่)[30][31]
ในระบบไอ-อี ของ E. coli Cas1 และ Cas 2 จับกันเป็นกลุ่มรวมโดยสองส่วน (dimer) ของ Cas2 จับเข้ากับสองส่วนของ Cas1[32] ในกลุ่มรวมนี้ Cas2 มีหน้าที่เป็นโครงสร้างแบบไม่ใช้เอนไซม์[32] และจับกับชิ้นสายคู่ของดีเอ็นเอจากภายนอก ขณะที่ Cas1 จับกับด้านข้างของดีเอ็เอส่วนสายเดี่ยวและช่วยเร่งในการรวมเข้ากับแถวลำดับคริสเปอร์[33][34][35] สเปสเซอร์ใหม่ถูกเพิ่มในส่วนเริ่มของคริสเปอร์ต่อจากลำดับนำ (leader sequence) ทำให้เป็นการบันทึกการติดเชื้อไวรัสตามลำดับเวลา[36]
โมทีฟติดกับโปรโตสเปสเซอร์
[แก้]การวิเคราะห์ทางชีวสารสนเทศศาสตร์บริเวณจีโนมเฟจที่ถูกตัดเป็นสเปสเซอร์ (เรียกว่าโปรโตสเปสเซอร์, protospacer) ชี้ว่าไม่ได้เป็นการเลือกอย่างสุ่ม ทว่าถูกพบติดกับลำดับดีเอ็นเอสั้น ๆ (3 – 5 คู่เบส) ที่เรียกว่า protospacer adjacent motifs หรือ แพม (PAM) การวิเคราะห์ของระบบคริสเปอร์-แคสแสดงว่าลำดับแพมมีความสำคัญสำหรับระบบแบบที่ 1 (type I) และแบบที่ 2 (type II) ทว่าไม่สำคัญในแบบที่ 3 (type III) ระหว่างการได้มาซึ่งสเปสเซอร์[37][38][39][40][41][42] ในระบบแบบที่ 1 และ 2 โปรโตสเปสเซอร์ถูกตัดบนตำแหน่งติดกับลำดับแพม โดยอีกด้านหนึ่งของสเปสเซอร์ถูกตัดโดย ruler mechanism ทำให้ขนาดของสเปสเซอร์ใกล้เคียงกันในแถวลำดับคริสเปอร์[43][44] การอนุรักษ์ลำดับแพมแตกต่างกันไปในคริสเปอร์-แคสแต่ละระบบและอาจเชื่อมโยงกับ Cas1 และลำดับนำ (leader sequence)[42][45]
สเปสเซอร์ใหม่ถูกเพิ่มเข้าไปในแถวลำดับคริสเปอร์โดยมีทิศทางที่แน่นอน[46] มักเลือกที่จะ[47][38][39][48][49] แต่ไม่เพียงแต่[41][44] ติดกับลำดับนำ การวิเคราห์ของระบบไอ-อีจาก E. coli แสดงว่าลำดับซ้ำแรกติดกับลำดับนำถูกคัดลอก โดยมี สเปสเซอร์ใหม่ที่พึ่งได้มาแทรกระหว่างลำดับซ้ำแรกและสอง[23][43]
ลำดับแพมน่าจะมีความสำคัญระหว่างการแทรกสเปสเซอร์ในระบบไอ-อี ลำดับนั้นมีส่วนประกอบของนิวคลีโอไทด์ท้ายที่ถูกคงไว้ติดกับนิวคลีโอไทด์แรกของโปรโตสเปสเซอร์ นิวครีโอไทด์นี้กลายเป็นเบสสุดท้ายของลำดับซ้ำโดยตรง (direct repeat) ชิ้นแรก[24][50][51] สิ่งนี้ชี้ว่ากลไกการได้มาซึ่งสเปสเซอร์ทำให้เกิดส่วนยื่นสายเดี่ยวในตำแหน่งรองท้ายของลำดับซ้ำโดยตรงและลำดับแพมระหว่างการแทรกสเปสเซอร์ อย่างไรก็ตาม ไม่ใช่ทุกแบบของคริสเปอร์-แคสจะใช้กลไกนี้ ด้วยความที่แพมในสิ่งมีชีวิตอื่นไม่แสดงการคงไว้ของตำแหน่งสุดท้ายในระดับเท่ากัน[45] เป็นไปได้ว่าระบบเหล่านี้ทำให้เกิดปลายทู่ที่ไม่มีส่วนยื่นออกมาตรงส่วนท้ายของลำดับซ้ำโดยตรงและโปรโตสเปสเซอร์ระหว่างการได้มาซี่งสเปสเซอร์
การประยุกต์
[แก้]ภายใน พ.ศ. 2557 มีงานวิจัยกว่า 1,000 งานที่กล่าวถึงคริสเปอร์ถูกตีพิมพ์[52][53] เทคโนโลยีถูกใช้เพื่อหยุดยั้งการปฏิบัติงานของยีนในเซลล์ไลน์มนุษย์และในเซลล์, เพื่อศึกษา Candida albicans, เพื่อปรับแต่งยีสต์เพื่อผลิตเชื้อเพลิงชีวภาพ และเพื่อปรับแต่งพันธุกรรมของสายพันธุ์พืช[53] คริสเปอร์ยังสามารถถูกใช้เพื่อเปลี่ยนยุงให้ไม่สามารถถ่ายทอดโรค เช่น มาลาเรีย[54]
การประเมินคำอ้างใหม่สำหรับความสัมพันธ์ระหว่างยีนกับโรคบนฐานของคริสเปอร์นำไปสู้การค้นพบความผิดปกติที่อาจมีความสำคัญ[55]

พันธุวิศวกรรม
[แก้]การแก้ไขจีโนมโดยคริสเปอร์/แคสไนน์ใช้ระบบคริสเปอร์แบบที่ 2 โดยใช้ Cas9, crRNA, tracrRNA และอาจใช้ร่วมกับส่วนของต้นแบบการซ่อมแซมดีเอ็นเอเพื่อช่วยในการเชื่อมต่อชิ้นส่วนโครโมโซมที่ไม่ใช่คู่ของมันเอง (non-homologous end joining, NHEJ) หรือ homology directed repair (HDR) ในการแก้ไขจีโนม

ส่วนประกอบหลัก
[แก้]| ส่วรประกอบ | หน้าที่ |
|---|---|
| crRNA | มีไกด์อาร์เอ็นเอซึ่งหาที่ตั้งของดีเอ็นเอของโฮสต์พร้อมกับตำแหน่ที่จับกับ tracrRNA (มักอยู่ในรูปห่วง, hairpin loop) เกิดเป็นกลุ่มรวมพร้อมปฏิบัติการ |
| tracrRNA | จับกับ crRNA และเกิดเป็นกลุ่มรวมพร้อมปฏิบัติการ |
| sgRNA | ย่อมาจาก Single guide RNAs เป็นการรวมกันของอาร์เอ็นเอประกอบด้วย tracrRNA และ crRNA อย่างน้อยหนึ่งชิ้น |
| Cas9 | โปรตีนที่รูปแบบพร้อมปฏิบัติการสามารถปรับแต่งดีเอ็นเอได้ มีหลายรูปแบบที่มีหน้าที่แตกต่างกัน (เช่น การตัดสายเดี่ยว การตัดสายคู่ การจับกับดีเอ็นเอ) เป็นผลจากหน้าที่ของ Cas9 ในการรู้จำตำแหน่งดีเอ็นเอ |
| ต้นแบบการซ่อมแซม (repair template) | ดีเอ็นเอที่นำกระบวนการซ่อมแซมเซลทำให้สามารถแทรกลำดับดีเอ็นเอเฉพาะได้ |
คริสเปอร์/แคสไนน์มักใช้พลาสมิดเพื่อบุกรุก (transfect) เซลล์เป้าหมาย[58] ส่วนประกอบหลักของพลาสมิดแสดงอยู่ในรูปทางขวา เริ่มจากการออกแบบ crRNA สำหรับการประยุกต์ใช้แต่ละครั้งด้วยความที่สิ่งนี้เป็นลำดับที่ Cas9 ใช้เพื่อระบุและจับโดยตรงกับดีเอ็นเอของเซลล์ crDNA ต้องจับกับที่ซึ่งต้องการแก้ไขเท่านั้น ต้นแบบการซ่อมแซมถูกออกแบบสำหรับการใช้แต่ละครั้งด้วยความที่ต้องทับซ้อนกับด้านใดด้านหนึ่งที่ถูกตัดและต้องเป็นรหัสสำหรับการแทรกลำดับ
crRNAs และ tracrRNA หลายชิ้นสามารถบรรจุเข้าด้วยกันเพื่อสร้าง single-guide RNA (sgRNA)[59] โดยสามารถนำ sgRNA นี้ไปต่อกับยีน Cas9 ในพลาสมิดเพื่อบุกรุกเข้าสู่เซลล์

อ้างอิง
[แก้]- ↑ Horvath P, Barrangou R (January 2010). "CRISPR/Cas, the immune system of bacteria and archaea". Science. 327 (5962): 167–70. Bibcode:2010Sci...327..167H. doi:10.1126/Science.1179555. PMID 20056882.
- ↑ Marraffini LA, Sontheimer EJ (March 2010). "CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea". Nature Reviews Genetics. 11 (3): 181–90. doi:10.1038/nrg2749. PMC 2928866. PMID 20125085.
- ↑ Barrangou R (2015). "The roles of CRISPR-Cas systems in adaptive immunity and beyond". Current Opinion in Immunology. 32: 36–41. doi:10.1016/j.coi.2014.12.008. PMID 25574773.
- ↑ Zhang F, Wen Y, Guo X (2014). "CRISPR/Cas9 for genome editing: progress, implications and challenges". Human Molecular Genetics. 23 (R1): R40–6. doi:10.1093/hmg/ddu125. PMID 24651067.
- ↑ Redman M, King A, Watson C, King D (August 2016). "What is CRISPR/Cas9?". Archives of Disease in Childhood. Education and Practice Edition. 101 (4): 213–5. doi:10.1136/archdischild-2016-310459. PMC 4975809. PMID 27059283.
- ↑ Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, และคณะ (March 2007). "CRISPR provides acquired resistance against viruses in prokaryotes". Science. 315 (5819): 1709–12. Bibcode:2007Sci...315.1709B. doi:10.1126/science.1138140. PMID 17379808. (ต้องลงทะเบียน)
- ↑ Marraffini LA, Sontheimer EJ (December 2008). "CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA". Science. 322 (5909): 1843–5. Bibcode:2008Sci...322.1843M. doi:10.1126/science.1165771. PMC 2695655. PMID 19095942.
- ↑ Mohanraju P, Makarova KS, Zetsche B, Zhang F, Koonin EV, van der Oost J (2016). "Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems". Science. 353 (6299): aad5147. doi:10.1126/science.aad5147. PMID 27493190.
- ↑ Grissa I, Vergnaud G, Pourcel C (May 2007). "The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats". BMC Bioinformatics. 8: 172. doi:10.1186/1471-2105-8-172. PMC 1892036. PMID 17521438.
- ↑ Sawyer E (9 February 2013). "Editing Genomes with the Bacterial Immune System". Scitable. Nature Publishing Group. สืบค้นเมื่อ 6 April 2015.
- ↑ Marraffini LA, Sontheimer EJ (March 2010). "CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea". Nature Reviews Genetics. 11 (3): 181–90. doi:10.1038/nrg2749. PMC 2928866. PMID 20125085.
- ↑ Ledford H (2015). "CRISPR, the disruptor". Nature. 522 (7554): 20–4. Bibcode:2015Natur.522...20L. doi:10.1038/522020a. PMID 26040877.
- ↑ 13.0 13.1 Snyder B (21 August 2014). "New technique accelerates genome editing process". research news @ Vanderbilt. Nashville, Tennessee: Vanderbilt University.
- ↑ Hendel A, Bak RO, Clark JT, Kennedy AB, Ryan DE, Roy S, Steinfeld I, Lunstad BD, Kaiser RJ, Wilkens AB, Bacchetta R, Tsalenko A, Dellinger D, Bruhn L, Porteus MH (September 2015). "Chemically modified guide RNAs enhance CRISPR-Cas genome editing in human primary cells". Nature Biotechnology. 33 (9): 985–9. doi:10.1038/nbt.3290. PMC 4729442. PMID 26121415.
- ↑ Ledford H (March 2016). "CRISPR: gene editing is just the beginning". Nature. 531 (7593): 156–9. doi:10.1038/531156a. PMID 26961639.
- ↑ Maxmen A (August 2015). "The Genesis Engine". WIRED. สืบค้นเมื่อ 2016-06-05.
- ↑ Travis J (17 December 2015). "Breakthrough of the Year: CRISPR makes the cut". Science Magazine. American Association for the Advancement of Science.
- ↑ Ledford H (June 2015). "CRISPR, the disruptor". Nature. 522 (7554): 20–4. doi:10.1038/522020a. PMID 26040877.
- ↑ Marraffini LA (October 2015). "CRISPR-Cas immunity in prokaryotes". Nature. 526 (7571): 55–61. doi:10.1038/nature15386. PMID 26432244.
- ↑ Aliyari R, Ding SW (January 2009). "RNA-based viral immunity initiated by the Dicer family of host immune receptors". Immunological Reviews. 227 (1): 176–88. doi:10.1111/j.1600-065X.2008.00722.x. PMC 2676720. PMID 19120484.
- ↑ Dugar G, Herbig A, Förstner KU, Heidrich N, Reinhardt R, Nieselt K, Sharma CM (May 2013). "High-resolution transcriptome maps reveal strain-specific regulatory features of multiple Campylobacter jejuni isolates". PLoS Genetics. 9 (5): e1003495. doi:10.1371/journal.pgen.1003495. PMC 3656092. PMID 23696746.
- ↑ Hatoum-Aslan A, Maniv I, Marraffini LA (December 2011). "Mature clustered, regularly interspaced, short palindromic repeats RNA (crRNA) length is measured by a ruler mechanism anchored at the precursor processing site". Proceedings of the National Academy of Sciences of the United States of America. 108 (52): 21218–22. Bibcode:2011PNAS..10821218H. doi:10.1073/pnas.1112832108. PMC 3248500. PMID 22160698.
- ↑ 23.0 23.1 Yosef I, Goren MG, Qimron U (July 2012). "Proteins and DNA elements essential for the CRISPR adaptation process in Escherichia coli". Nucleic Acids Research. 40 (12): 5569–76. doi:10.1093/nar/gks216. PMC 3384332. PMID 22402487.
- ↑ 24.0 24.1 Swarts DC, Mosterd C, van Passel MW, Brouns SJ (2012). "CRISPR interference directs strand specific spacer acquisition". PLoS One. 7 (4): e35888. Bibcode:2012PLoSO...735888S. doi:10.1371/journal.pone.0035888. PMC 3338789. PMID 22558257.
- ↑ Babu M, Beloglazova N, Flick R, Graham C, Skarina T, Nocek B, และคณะ (January 2011). "A dual function of the CRISPR-Cas system in bacterial antivirus immunity and DNA repair". Molecular Microbiology. 79 (2): 484–502. doi:10.1111/j.1365-2958.2010.07465.x. PMC 3071548. PMID 21219465.
- ↑ Han D, Lehmann K, Krauss G (June 2009). "SSO1450--a CAS1 protein from Sulfolobus solfataricus P2 with high affinity for RNA and DNA". FEBS Letters. 583 (12): 1928–32. doi:10.1016/j.febslet.2009.04.047. PMID 19427858.
- ↑ Wiedenheft B, Zhou K, Jinek M, Coyle SM, Ma W, Doudna JA (June 2009). "Structural basis for DNase activity of a conserved protein implicated in CRISPR-mediated genome defense". Structure. 17 (6): 904–12. doi:10.1016/j.str.2009.03.019. PMID 19523907.
- ↑ Wiedenheft B, Sternberg SH, Doudna JA (February 2012). "RNA-guided genetic silencing systems in bacteria and archaea". Nature. 482 (7385): 331–8. Bibcode:2012Natur.482..331W. doi:10.1038/nature10886. PMID 22337052.
- ↑ Beloglazova N, Brown G, Zimmerman MD, Proudfoot M, Makarova KS, Kudritska M, และคณะ (July 2008). "A novel family of sequence-specific endoribonucleases associated with the clustered regularly interspaced short palindromic repeats". The Journal of Biological Chemistry. 283 (29): 20361–71. doi:10.1074/jbc.M803225200. PMC 2459268. PMID 18482976.
- ↑ Samai P, Smith P, Shuman S (December 2010). "Structure of a CRISPR-associated protein Cas2 from Desulfovibrio vulgaris". Acta Crystallographica Section F. 66 (Pt 12): 1552–6. doi:10.1107/S1744309110039801. PMC 2998353. PMID 21139194.
- ↑ Nam KH, Ding F, Haitjema C, Huang Q, DeLisa MP, Ke A (October 2012). "Double-stranded endonuclease activity in Bacillus halodurans clustered regularly interspaced short palindromic repeats (CRISPR) -associated Cas2 protein". The Journal of Biological Chemistry. 287 (43): 35943–52. doi:10.1074/jbc.M112.382598. PMC 3476262. PMID 22942283.
- ↑ 32.0 32.1 Nuñez JK, Kranzusch PJ, Noeske J, Wright AV, Davies CW, Doudna JA (June 2014). "Cas1-Cas2 complex formation mediates spacer acquisition during CRISPR-Cas adaptive immunity". Nature Structural & Molecular Biology. 21 (6): 528–34. doi:10.1038/nsmb.2820. PMC 4075942. PMID 24793649.
- ↑ Nuñez JK, Lee AS, Engelman A, Doudna JA (March 2015). "Integrase-mediated spacer acquisition during CRISPR-Cas adaptive immunity". Nature. 519 (7542): 193–8. doi:10.1038/nature14237. PMC 4359072. PMID 25707795.
- ↑ Wang J, Li J, Zhao H, Sheng G, Wang M, Yin M, Wang Y (November 2015). "Structural and Mechanistic Basis of PAM-Dependent Spacer Acquisition in CRISPR-Cas Systems". Cell. 163 (4): 840–53. doi:10.1016/j.cell.2015.10.008. PMID 26478180.
- ↑ Nuñez JK, Harrington LB, Kranzusch PJ, Engelman AN, Doudna JA (November 2015). "Foreign DNA capture during CRISPR-Cas adaptive immunity". Nature. 527 (7579): 535–8. doi:10.1038/nature15760. PMC 4662619. PMID 26503043.
- ↑ Sorek R, Lawrence CM, Wiedenheft B (2013). "CRISPR-mediated adaptive immune systems in bacteria and archaea". Annual Review of Biochemistry. 82 (1): 237–66. doi:10.1146/annurev-biochem-072911-172315. PMID 23495939.
- ↑ Bolotin A, Quinquis B, Sorokin A, Ehrlich SD (August 2005). "Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin". Microbiology. 151 (Pt 8): 2551–61. doi:10.1099/mic.0.28048-0. PMID 16079334.
- ↑ 38.0 38.1 Horvath P, Romero DA, Coûté-Monvoisin AC, Richards M, Deveau H, Moineau S, และคณะ (February 2008). "Diversity, activity, and evolution of CRISPR loci in Streptococcus thermophilus". Journal of Bacteriology. 190 (4): 1401–12. doi:10.1128/JB.01415-07. PMC 2238196. PMID 18065539.
- ↑ 39.0 39.1 Deveau H, Barrangou R, Garneau JE, Labonté J, Fremaux C, Boyaval P, Romero DA, Horvath P, Moineau S (February 2008). "Phage response to CRISPR-encoded resistance in Streptococcus thermophilus". Journal of Bacteriology. 190 (4): 1390–400. doi:10.1128/JB.01412-07. PMC 2238228. PMID 18065545.
- ↑ Mojica FJ, Díez-Villaseñor C, García-Martínez J, Almendros C (March 2009). "Short motif sequences determine the targets of the prokaryotic CRISPR defence system". Microbiology. 155 (Pt 3): 733–40. doi:10.1099/mic.0.023960-0. PMID 19246744.
- ↑ 41.0 41.1 Lillestøl RK, Shah SA, Brügger K, Redder P, Phan H, Christiansen J, Garrett RA (April 2009). "CRISPR families of the crenarchaeal genus Sulfolobus: bidirectional transcription and dynamic properties". Molecular Microbiology. 72 (1): 259–72. doi:10.1111/j.1365-2958.2009.06641.x. PMID 19239620.
- ↑ 42.0 42.1 Shah SA, Hansen NR, Garrett RA (February 2009). "Distribution of CRISPR spacer matches in viruses and plasmids of crenarchaeal acidothermophiles and implications for their inhibitory mechanism". Biochemical Society Transactions. 37 (Pt 1): 23–8. doi:10.1042/BST0370023. PMID 19143596.
- ↑ 43.0 43.1 Díez-Villaseñor C, Guzmán NM, Almendros C, García-Martínez J, Mojica FJ (May 2013). "CRISPR-spacer integration reporter plasmids reveal distinct genuine acquisition specificities among CRISPR-Cas I-E variants of Escherichia coli". RNA Biology. 10 (5): 792–802. doi:10.4161/rna.24023. PMC 3737337. PMID 23445770.
- ↑ 44.0 44.1 Erdmann S, Garrett RA (September 2012). "Selective and hyperactive uptake of foreign DNA by adaptive immune systems of an archaeon via two distinct mechanisms". Molecular Microbiology. 85 (6): 1044–56. doi:10.1111/j.1365-2958.2012.08171.x. PMC 3468723. PMID 22834906.
- ↑ 45.0 45.1 Shah SA, Erdmann S, Mojica FJ, Garrett RA (May 2013). "Protospacer recognition motifs: mixed identities and functional diversity". RNA Biology. 10 (5): 891–9. doi:10.4161/rna.23764. PMC 3737346. PMID 23403393.
- ↑ Pourcel C, Salvignol G, Vergnaud G (March 2005). "CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies". Microbiology. 151 (Pt 3): 653–63. doi:10.1099/mic.0.27437-0. PMID 15758212.
- ↑ Tyson GW, Banfield JF (January 2008). "Rapidly evolving CRISPRs implicated in acquired resistance of microorganisms to viruses". Environmental Microbiology. 10 (1): 200–7. doi:10.1111/j.1462-2920.2007.01444.x. PMID 17894817.
- ↑ Andersson AF, Banfield JF (May 2008). "Virus population dynamics and acquired virus resistance in natural microbial communities". Science. 320 (5879): 1047–50. Bibcode:2008Sci...320.1047A. doi:10.1126/science.1157358. PMID 18497291.
- ↑ Pride DT, Sun CL, Salzman J, Rao N, Loomer P, Armitage GC, และคณะ (January 2011). "Analysis of streptococcal CRISPRs from human saliva reveals substantial sequence diversity within and between subjects over time". Genome Research. 21 (1): 126–36. doi:10.1101/gr.111732.110. PMC 3012920. PMID 21149389.
- ↑ Goren MG, Yosef I, Auster O, Qimron U (October 2012). "Experimental definition of a clustered regularly interspaced short palindromic duplicon in Escherichia coli". Journal of Molecular Biology. 423 (1): 14–6. doi:10.1016/j.jmb.2012.06.037. PMID 22771574.
- ↑ Datsenko KA, Pougach K, Tikhonov A, Wanner BL, Severinov K, Semenova E (July 2012). "Molecular memory of prior infections activates the CRISPR/Cas adaptive bacterial immunity system". Nature Communications. 3: 945. Bibcode:2012NatCo...3E.945D. doi:10.1038/ncomms1937. PMID 22781758.
- ↑ Doudna JA, Charpentier E (November 2014). "Genome editing. The new frontier of genome engineering with CRISPR-Cas9". Science. 346 (6213): 1258096. doi:10.1126/science.1258096. PMID 25430774.
- ↑ 53.0 53.1 Ledford H (June 2015). "CRISPR, the disruptor". Nature. 522 (7554): 20–4. Bibcode:2015Natur.522...20L. doi:10.1038/522020a. PMID 26040877.
- ↑ Alphey L (2016). "Can CRISPR-Cas9 gene drives curb malaria?". Nature Biotechnology. 34 (2): 149–50. doi:10.1038/nbt.3473. PMID 26849518.
- ↑ Ledford H (2017). "CRISPR studies muddy results of older gene research". Nature. doi:10.1038/nature.2017.21763.
- ↑ "CRISPR/Cas9 Plasmids". www.systembio.com. คลังข้อมูลเก่าเก็บจากแหล่งเดิมเมื่อ 2015-12-22. สืบค้นเมื่อ 2015-12-17.
- ↑ "CRISPR Cas9 Genome Editing". www.origene.com. OriGene. สืบค้นเมื่อ 2015-12-17.
- ↑ 58.0 58.1 Ran FA, Hsu PD, Wright J, Agarwala V, Scott DA, Zhang F (November 2013). "Genome engineering using the CRISPR-Cas9 system". Nature Protocols. 8 (11): 2281–308. doi:10.1038/nprot.2013.143. PMC 3969860. PMID 24157548.
- ↑ Ly, Joseph (2013). Discovering Genes Responsible for Kidney Diseases (Ph.D.). University of Toronto. สืบค้นเมื่อ 26 December 2016.
ดูเพิ่ม
[แก้]- Doudna J, Mali P (23 March 2016). CRISPR-Cas: A Laboratory Manual. New York: Cold Spring Harbor Laboratory Press. ISBN 978-1-62182-131-1.
- Mohanraju P, Makarova KS, Zetsche B, Zhang F, Koonin EV, van der Oost J (August 2016). "Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems". Science. 353 (6299): aad5147. doi:10.1126/science.aad5147. PMID 27493190.
- Sander JD, Joung JK (April 2014). "CRISPR-Cas systems for editing, regulating and targeting genomes". Nature Biotechnology. 32 (4): 347–55. doi:10.1038/nbt.2842. PMC 4022601. PMID 24584096.
- Slaymaker IM, Gao L, Zetsche B, Scott DA, Yan WX, Zhang F (January 2016). "Rationally engineered Cas9 nucleases with improved specificity". Science. 351 (6268): 84–8. doi:10.1126/science.aad5227. PMC 4714946. PMID 26628643.
- Terns RM, Terns MP (March 2014). "CRISPR-based technologies: prokaryotic defense weapons repurposed". Trends in Genetics. 30 (3): 111–8. doi:10.1016/j.tig.2014.01.003. PMC 3981743. PMID 24555991.
- Westra ER, Buckling A, Fineran PC (May 2014). "CRISPR-Cas systems: beyond adaptive immunity". Nature Reviews Microbiology. 12 (5): 317–26. doi:10.1038/nrmicro3241. PMID 24704746.
- Andersson AF, Banfield JF (May 2008). "Virus population dynamics and acquired virus resistance in natural microbial communities". Science. 320 (5879): 1047–50. Bibcode:2008Sci...320.1047A. doi:10.1126/science.1157358. PMID 18497291.
- Hale C, Kleppe K, Terns RM, Terns MP (December 2008). "Prokaryotic silencing (psi) RNAs in Pyrococcus furiosus". RNA. 14 (12): 2572–9. doi:10.1261/rna.1246808. PMC 2590957. PMID 18971321.
- van der Ploeg JR (June 2009). "Analysis of CRISPR in Streptococcus mutans suggests frequent occurrence of acquired immunity against infection by M102-like bacteriophages". Microbiology. 155 (Pt 6): 1966–76. doi:10.1099/mic.0.027508-0. PMID 19383692.
- van der Oost J, Brouns SJ (November 2009). "RNAi: prokaryotes get in on the act". Cell. 139 (5): 863–5. doi:10.1016/j.cell.2009.11.018. PMID 19945373.
- Karginov FV, Hannon GJ (January 2010). "The CRISPR system: small RNA-guided defense in bacteria and archaea". Molecular Cell. 37 (1): 7–19. doi:10.1016/j.molcel.2009.12.033. PMC 2819186. PMID 20129051.
- Pul U, Wurm R, Arslan Z, Geissen R, Hofmann N, Wagner R (March 2010). "Identification and characterization of E. coli CRISPR-cas promoters and their silencing by H-NS". Molecular Microbiology. 75 (6): 1495–512. doi:10.1111/j.1365-2958.2010.07073.x. PMID 20132443.
- Díez-Villaseñor C, Almendros C, García-Martínez J, Mojica FJ (May 2010). "Diversity of CRISPR loci in Escherichia coli". Microbiology. 156 (Pt 5): 1351–61. doi:10.1099/mic.0.036046-0. PMID 20133361.
- Deveau H, Garneau JE, Moineau S (2010). "CRISPR/Cas system and its role in phage-bacteria interactions". Annual Review of Microbiology. 64: 475–93. doi:10.1146/annurev.micro.112408.134123. PMID 20528693.
- Koonin EV, Makarova KS (December 2009). "CRISPR-Cas: an adaptive immunity system in prokaryotes". F1000 Biology Reports. 1: 95. doi:10.3410/B1-95. PMC 2884157. PMID 20556198.
- "The age of the red pen". The Economist. August 22, 2015. ISSN 0013-0613. สืบค้นเมื่อ 2015-08-25.
